Edição do Genoma das Plantas
29 de outubro de 2019
Alejandro Hernández, Diretor de biotecnologia para a América Central e o Caribe da CropLife Latin America
A edição do genoma nas plantas é alcançada graças à biotecnologia de precisão e visa ser um marco significativo na agricultura moderna. Atualmente, existem pedidos de permissão para vender tomates, arroz, milho, trigo, soja e cogumelos nos Estados Unidos. A edição genética promete mudanças nutricionais, como a produção de trigo sem glúten ou a redução de gorduras trans nos óleos de soja. Em que consiste?
O genoma é todo o material genético de um organismo ou de uma determinada espécie. No caso de organismos superiores ou eucarióticos, como plantas e animais, existe o genoma nuclear e o genoma de plastídios (plantas) e/ou mitocôndrias (animais e plantas).
O material genético é apresentado em estruturas altamente organizadas no DNA e é um código genético escrito em quatro letras: A (adenina) que combina com T (timina) e C (citosina) que combina com G (guanina).

A sequência de um genoma vegetal é semelhante a uma biblioteca de letras. Dentro desta biblioteca de letras, há uma pequena proporção que corresponde a genes ou sequências com alguma função. Esses genes têm pequenas alterações que podem variar sua função. No melhoramento genético convencional, essas mudanças são as características procuradas pela polinização ou por mudanças aleatórias. Até poucas décadas atrás, sabíamos muito pouco sobre genomas e muito menos sobre como ajustá-los, melhorá-los ou repará-los quando encontramos um erro. Hoje, as ferramentas de sequenciamento e as novas técnicas de Edição de Genomas possibilitam fazer e verificar com extrema precisão os ajustes no material genético. No caso das plantas, o cenário é muito promissor, pois você pode obter os mesmos resultados que no melhoramento convencional, mas em um tempo muito menor. Portanto, eles são chamados de novas técnicas de melhoramento genético vegetal, em inglês New Breeding Techniques/ Technologies (NBTs).
As tecnologias de edição de genomas são baseadas em 2 princípios. O primeiro é que você deve localizar o local exato no genoma para poder executar alguma função. O mais comum, mas não a única, é cortar a fita dupla do DNA (em inglês "double-strand breaks" (DSBs)). O segundo princípio é o uso de mecanismos naturais de reparo do genoma. Nos organismos superiores ou eucarióticos, é chamado reparo homólogo quando uma cópia de backup idêntica é usada para reparar o genoma; ou não homólogo quando o genoma é reparado sem a ajuda de um fio de reparo. O resultado final é uma sequência de DNA com reparos ou ajustes, que na literatura foram originalmente chamados de técnicas “SDN” ou “SSN” do inglês “site directed nucleases”, ou “site specific nucleases” 1, 2 y 3.

Vamos explorar esses dois conceitos para entender o estado da arte das novas técnicas de edição de genomas.
Princípio 1. Localize o lugar certo no genoma
Os primeiros métodos para identificar algum material genético foram descobertos nos anos 80 como mecanismos de defesa contra vírus. Imagine uma tesoura de DNA, que na época era chamada de endonucleases e meganucleases, eles tinham a capacidade de reconhecer cerca de 6 a 8 letras as primeiras e 12-40 letras as segundas, e podiam fazer um corte no DNA. Nos anos 90 e início de 2000, houve progresso na geração de nucleases de dedo de zinco (reconhecer 6-18 pares de bases) e as TALENS (Transcription-Activator-Like Effector Nucleases) que reconhecem 12-31 pb. Enquanto que, de 2012 até hoje, uma série de tesouras ou enzimas moleculares chamadas CAS foram identificadas que estão associadas a bibliotecas de pequenos fragmentos de aproximadamente 20 pares de bases de sequências de vírus denominadas CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). Estes são os chamados CRISPR-CAS9/ CAS12a, CAS12b, CAS12e, CAS13a, CAS13b, CAS14.
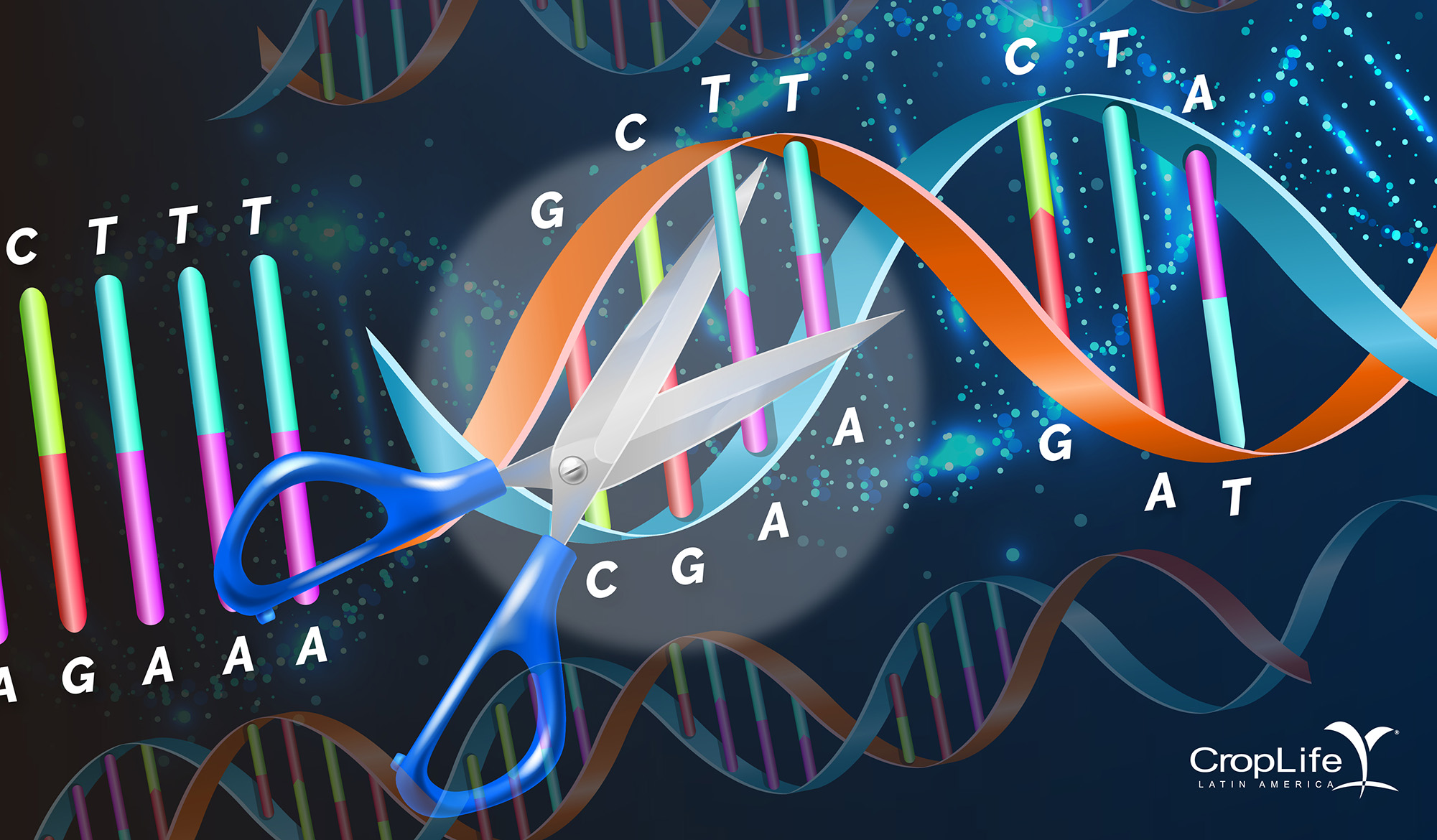
A história do CRISPR-CAS9 é cativante
A enzima foi descoberta por Francisco Mojica em 1993 quando ele estudou o microorganismo Haloferax mediterranei e notou um sistema de defesa de bactérias contra vírus composto por dois elementos, uma tesoura chamada CAS e uma biblioteca de informações que usava a tesoura para identificar o vírus. Este último chamou CRISPR em referência à serie de sequências de vírus que foram muito ordenadas uma após a outra e separadas por um DNA idêntico que foi repetido várias vezes, daí o acrônimo “Clustered Regularly Interspaced Short Palindromic Repeats”.

A descoberta passou por muitos anos com baixo perfil, até 2012, quando foi verificado que o sistema bacteriano poderia ser usado para editar o DNA genômico. A seguir, aparece no cenário internacional uma competição científica no mais alto nível entre duas universidades nos Estados Unidos: a Universidade de Berkeley na costa oeste sob o comando dos dois cientistas Jennifer Doudna e Emmanuelle Charpentier[1] e o Harvard-MIT Broad Institute na costa leste liderado pelo cientista Feng Zhang. A descoberta de ambos os grupos foi baseada no fato de que o CAS9 (a tesoura) é perfeitamente complementado por uma sequência que poderia ser projetada sob medida chamada RNA guia, que contém 20 letras usadas como referência para localizar um local específico no genoma, tornando-a extremamente específica.
Princípio 2, reparo do genoma
Depois que um corte na fita dupla do DNA é feito, o mecanismo de reparo natural da célula entra em ação. A maquinaria celular pode reparar o genoma de maneiras diferentes e, dependendo do tipo de reparo, é chamado de “SDN” ou “SSN” em inglês “site directed nucleases”, ou “site specific nucleases” 1, 2 y 3.
SDN ou SSN-1 são produtos da recombinação não homóloga, ou seja, um reparo que não utiliza um fio de reparo e resulta em inserções, deleções ou pequenas alterações.
SDS-2 usa um DNA de fita e, portanto, o reparo é homólogo, mas não insere um novo DNA, mas repara a mensagem; por exemplo, pode causar alterações no nível dos aminoácidos da proteína que codifica.
Recursos
1. Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., & Charpentier, E. (2012). A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. science, 337(6096), 816-821.

















